














Prepara tus exámenes y mejora tus resultados gracias a la gran cantidad de recursos disponibles en Docsity

Gana puntos ayudando a otros estudiantes o consíguelos activando un Plan Premium


Prepara tus exámenes
Prepara tus exámenes y mejora tus resultados gracias a la gran cantidad de recursos disponibles en Docsity
Prepara tus exámenes con los documentos que comparten otros estudiantes como tú en Docsity
Los mejores documentos en venta realizados por estudiantes que han terminado sus estudios
Estudia con lecciones y exámenes resueltos basados en los programas académicos de las mejores universidades
Responde a preguntas de exámenes reales y pon a prueba tu preparación

Consigue puntos base para descargar
Gana puntos ayudando a otros estudiantes o consíguelos activando un Plan Premium
Comunidad
Pide ayuda a la comunidad y resuelve tus dudas de estudio
Descubre las mejores universidades de tu país según los usuarios de Docsity
Ebooks gratuitos
Descarga nuestras guías gratuitas sobre técnicas de estudio, métodos para controlar la ansiedad y consejos para la tesis preparadas por los tutores de Docsity
Hola, hice estos esquemas que resumen la parte de biología molecular del Karp, espero les ayude :')
Tipo: Esquemas y mapas conceptuales
1 / 24

Esta página no es visible en la vista previa
¡No te pierdas las partes importantes!

















La célula es la unidad estructural y funcional de la vida Todos los organismos celulares poseen ADN Son organismos que pueden vivir independientemente Todas las células poseen membrana plasmática Lenguaje genético idéntico Todas poseen citoplasma Existen dos tipos principales:
1era clase Eucariota pro- antes, karion- núcleo Eu- verdadero, karion- núcleo Su núcleo, llamado nucleoide, está en contacto directo con el citoplasma al igual que sus orgánulos Su núcleo está envuelto por una membrana al igual que sus organelos que los separa del citoplasma Su tipo de nutrición es heterotrofa Vacuolas de gas Su reproducción es por división binaria o parasexual No realizar endocitosis ni exocitosis Procariota Se dividen en dos grupos Su tipo de nutrición es autótrofa Poseen una pared celular y la membrana celular Se dividen en cuatro grupos Protozooarios Hongo Planta Animal Sus organelos son más complejos Existencia de microambientes ADN lineal ADN circular
Parásitos considerados seres vivos no considerado como células muchos en lugar de adn tienen arn Su forma de invadir la célula cambia según si es ADN o ARN ADN ARN Se introduce en la célula y se dirige al núcleo al cual entra y se inserta en el ADN de la célula. El cuerpo la detecta cuando reconoce proteínas raras Con su enzima retrotranscriptasa inversa, se auto-reescribe el arnm pero cambia el uracilo por timina y la ribosa por desoxirribosa 18/08/ 2da clase Biomoléculas Las biomoléculas son las moléculas que se encuentran presentes en todos los seres vivos. Están formadas por sustancias químicas compuestas principalmente por carbono, hidrógeno, oxígeno, nitrógeno, fósforo y azufre Se dividen en 4 grandes grupos
Fuente principal de energía Fórmula general de CH2O Soluble en disolventes orgánicos, ej cloroformo Su forma mínima es el ácido graso, Formado de aminoácidos Un aminoácido cuenta con un grupo amino (+) Formado a partir de nucleótidos Dos tipos: ácido desoxirribonucleico
Se pueden clasificar según: Su número de carbonos : 3 triosa, 4 tetrosa... Por posición: cetona-cetosa (fructuosa), aldehído-aldosa (glucosa) Por unidades: glucosa+ sacarosa= fructuosa Disacáridos : unión de dos monosacaridos por enlace glucosídico (entre C1 y el hidroxilo de otro azucar) Glucosa+glucosa= maltosa Oligosacaridos: unión de algunos monosacaridos, ej. glucocalix Polisacaridos : Nutricionales : glucógeno y almidón (plantas) Estructurales : celulosa, quitina compuesto de una fila de hidrocarburos (hidrofóbico) unido a grupo carboxílo (hidrofílico) Se pueden clasificar en tres grupos:
Una molécula de glicerol unida a tres ácidos grasos por medio de un enlace estér Los ácidos grasos pueden ser saturados (enlace sencillo) o insaturados (doble enlace)
y un grupo carboxílo (-) unidos por un carbono al que se le añade una cadena lateral Se unen por enlaces peptídicos Una vez incorporados a los aminoacidos se les llama residuos Una cadena promedio tiene 450 aminácidos, pero solo 20 diferentes se usan en su construcción Los aminoácidos en la sintesis de proteínas son siempre L- aminoácidos *El carbono central de todos los aminoácidos, excepto la glicina están unidos a cuatro grupos diferentes Ácido aspártico, ácido glutámico, lisina y Polar, con carga Cadenas laterales (o grupo R) Cadenas laterales (o grupo R) ( DNA ) y ácido ribonucleico ( RNA ) Nucleótido: -Un ázucar de 5 carbonos -Una base nitrogenada -Un grupo fosfato *El azúcar y la base forman un nucleósido El fosfato está ligado al carbono 5´del ázucar y la base nitrogenada está unida al carbono 1 del azúcar Existen 2 tipos de bases: Pirimidas : citosina, uracilo y timina (un solo anillo) Purinas : adenina y guanina (dos anillos)
Modificaciones postraducciona- les (PTM): Aminoácidos que surgen por alteraciones en las cadenas laterales después de su incorporación a una cadena polipeptídica Estructuras Primaria Secuencia lineal con 20 aminoa diferentes Secundaria Dos conformaciones: Hélice alfa: espiral con las cadenas laterales proyectadas afuera Lámina beta: la cadena principal adopta una forma plegada Se estabiliza princ. por enlaces de hidrógeno Terciaria -Determina las interacciones y actividad enzimática. -En las eu las proteínas se componen de dos o mas dominios -Las proteínas se pueden clasificar en Firbrosas (alargadas) o globulares (compactas, la mayoria dentro de la célula) -Se estabiliza princ. por enlaces no covalentes. Cuartenaria Prolina La mayoria se encuentra en complejos de más de una subunidad. Unidas por enlaces de disulfuro o en su mayoria por no covalentes Homodímero: dos
3ra clase
Procariotas (bacterias) Eucariotas Un solo oriC u origen de replicación donde está la DNA A (reconoce el origen) Varios orígenes de replicación unidos a la proteína ORC (complejo de reconocimiento de origen)
4ta clase *Horquilla de replicación = Zona del DNA donde se produce el desenrollamiento de las dos cadenas polinucleotídicas y la síntesis de las nuevas cadenas.
3er paso: final de la replicación DNA polimerasa I: Elimina los RNA primarios y llena los espacios con DNA. (en algunos bacterias existe un mecanismo llamado reparación de desapareamientos que opera después de la replicación) *DNA polimerasa I: Consiste en una sola unidad. Cuando se incorpora un nucleótido incorrecto por la dna polimerasa I, esta se paraliza y elimina el nucleótido mal apareado en su sitio exonucleasa (los DNA polimerasa bacterianas poseen actividad exonucleasa) Primasa: la iniciación se realiza con un RNA polimerasa, la primasa, que construye un primario corto de RNA (primaria) PCNA : abrazadera corrediza RCF : cargador de la abrazadera corrediza DNA ligasa : enzima que une los fragmentos de Okazaki replicación *Polimerasas: Polimerasa γ: replica el ADN mitocondrial Polimerasa B: reparación del ADN Polimerasa α : se asocia con la primasa y juntas inician la síntesis de los fragmentos de Okazaki Polimerasa δ: enzima primaria que sintetiza el DNA durante la preplicación de la hebra retrasada. Polimerasa ε: enzima primaria que sintetiza el DNA durante la preplicación de la hebra adelantada 3er paso: final de la replicación
FEN-1 (DNA poli 1) : elimina los RNA primarios
Bacterias Eucariotas *Todas las polimerasa sintetizan las hebras de DNA en dirección 5´-3´ añadiendo solo en el grupo hidroxilo, debido a esto se da la replicación de una hebra adelantada y otra atrasada 24 y 25 /08/ 5-6 clase
Caja pribnow o caja tata: l as bases en -10 tienen la secuencia TATAAT El nucleótido que inicia la transcripción se denota como +1 y el que le precede -1, laspartes del DNA que preceden el sitio de inicia están corriente arriba , y las que las suceden corriente abajo Enlongación Durante la elongación, la ARN polimerasa "camina" sobre la hebra del ADN, conocida como la hebra molde, en la dirección 3' a 5' (recordando que siempre agrega en 3´) Terminación Dependiente de RHO: rodea el RNA recién sintetizada y la separa del DNA Enlongación Factores de elongación: TFEb: cinasa que fosforila los residuos de serina en el C- terminal (carboxilo) de la polimerasa, esto estimula la elongación TF II S: ayuda a incrementar la velocidad de la transcripción *Una vez comenzada la trans algunos GTF se pueden dejar atrás o se liberan del complejo *La RNA polimerasa II tiene una estructura inusual (heptapéptidos) en el dominio carboxilo termina. La RNA polimerasa que se ensambla en el momento de pre inicio no está fosforilada, pero ya en la transcripción está fuertemente forforilada Terminación
Secuencias de terminación: existen en regiones (ricas en A y T) con repeticiones invertidas que pueden unirse y formar una horquilla de terminación, esta desvía a la polimerasa Capping 5´: en el extremo 5´se forma una tapa de metilguanosina Poliadenización 3´: al encontrarse con muchas bases adeninas las corta por factores de corte y la el extremo 3´ termina con una cola poliadenizada (marca el tiempo de vida del RNA)
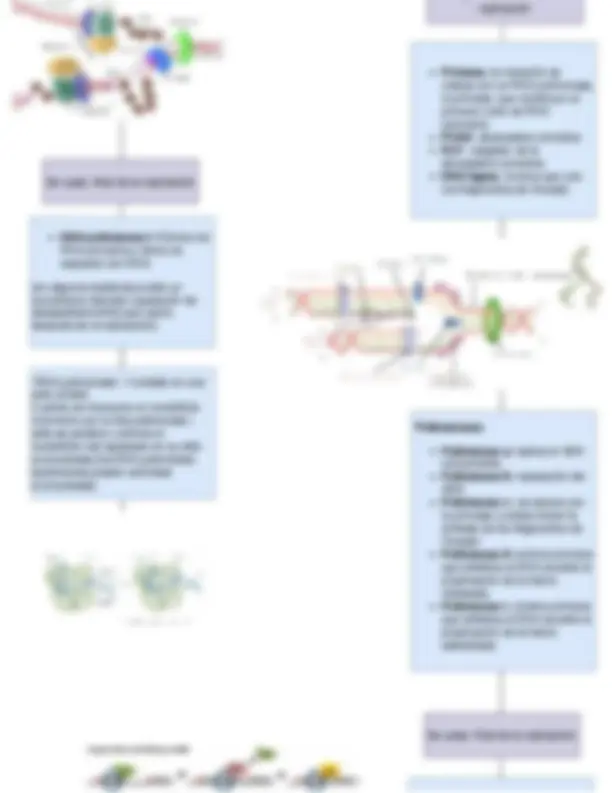
subunidad grande (50s) se una prematuramente a la pequeña (30s) Paso 2: traer el primer aa-tRNA AUG: es el único codón para metionina N-formilmetionina: siempre la metionina es el primer aminoácido que se incorpora al extremo N de la hebra de RNA pero en bacterias reacciona con un grupo formilo. El complejo 43s se recluta al mRNA con la ayuda de factores de inicio que ya están unidos al mRNA eIF4E : se une a la tapa 5´ eIF4A : se mueve a lo lardo de 5´eliminando cualquier región de doble hebra que pueda interferir con el movimiento de 40S eIF4G : sirve de conector entre el tapado 5´y el extremo 3´poliadenilado El escaneo sigue hasta que alcanza una secuencia reconocible que contiene el codón de inicio AUG. Una vez alcanzado el GTP unido a IF2 se hidroliza y la subunidad grande se une al complejo La formación del ribosoma 80S requiere de IF5B-GTP cuyo GTP tambien se hidroliza, se liberan los factores de inicio y pasa a la elongación
Paso 2: formación de enlaces peptídicos Se realiza cuando el nitrógeno amínico del RNAaa en el sitio A se une al carbono carbonílico del aminoácido del sitio P, desplazándolo. La formación de enlaces peptídicos ocurre sin la entrada de energía externa sino que la peptidil transferasa (una ribozima) cataliza la reacción Se caracteriza por un pequeño movimiento de trinquete de 6° de subuni pequeña Paso 3: translocación Como resultado el ribosoma se mueve 3 nucleótidos (5´-3´) y va acompañada del movimiento de: Dipeptidil-tRNA: desde el sitio A hasta el P (la ribozima) El tRNA desacilado: desde el P haste el E *Se ha descubierto que los tRNA ocupan estados híbridos A/P y P/E RF1 : reconoce los codones de terminación UAA y UAG RF2: reconoce los codones de terminación UAA y UGA
Una vez que se ha cambiado al estado híbrido un factor de enlongación GTP: EF-G : bacterias eEF2 : eucariotas se unen al ribosoma para evitar que se muevan los tRNA y vuelvan a la conformación clásica A/A o P/P Entonces el GTP ligado se hidroliza y coloca los tRNA en los estados E/E y P/P dejando el sitio A vacio Paso 4: liberación del tRNA desaciliado *Por cada ciclo de elongación dos GTP se hidrolizan, una durante la selección del aminoacil-tRNA y otra en la translocacion El tRNA desacilado sale del ribosoma y deja vacío el sitio E
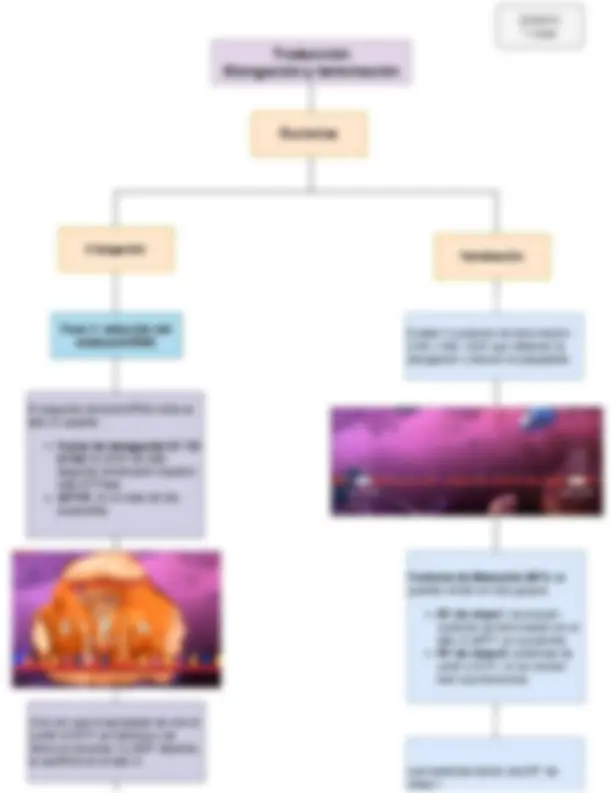
La RNA eucariótica requiere de la adicón de una tapa 5´y una cola 3´polia (A) y la eliminación de cualquier intrón intermedio, solo hasta que termina está listo para su exportación desde el núcleo